データセットの準備
library (randomForest) # データの概要 DV1 DV2 DV3 DV4 DV5 DV6 DV7 DV8 DV9 IV 1 0 0 4 4 4 5 5 5 5 4 2 0 0 4 5 5 5 5 5 5 5 3 0 0 4 4 4 5 3 4 4 5 4 0 0 4 5 4 3 5 5 5 5 5 0 0 5 4 2 5 5 5 5 5 6 0 0 4 4 4 4 4 4 4 4 # ランダムサンプリングの結果(乱数)を固定 set.seed (100) # データのx%を抽出しトレーニング・テストデータに分ける smpl <- sample(nrow(dat), 0.3*nrow(dat)) train <- dat[smpl, ] test <- dat[-smpl, ] # データセットを説明変数と予測変数に分ける features <- train[1 : 9] labels <- train[10] # 予測変数をファクターに変換する(カテゴリ変数の場合) labels <- as.factor(labels[[1]])
モデルを作成する
# Model Description rf.model <- randomForest(x = features, y = labels, importance = TRUE, proximity = TRUE)
結果の可視化
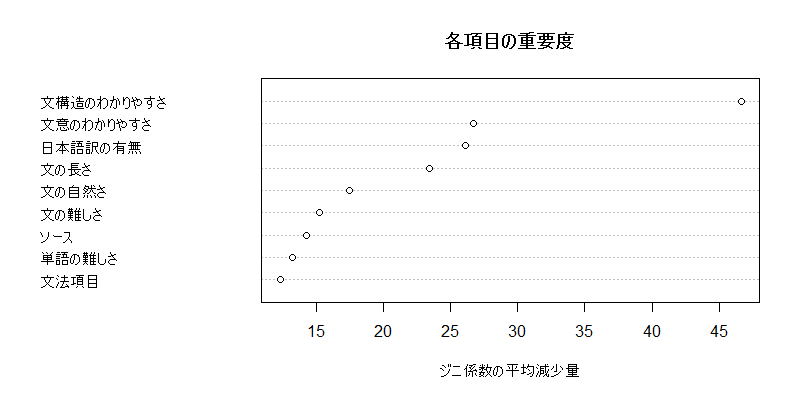
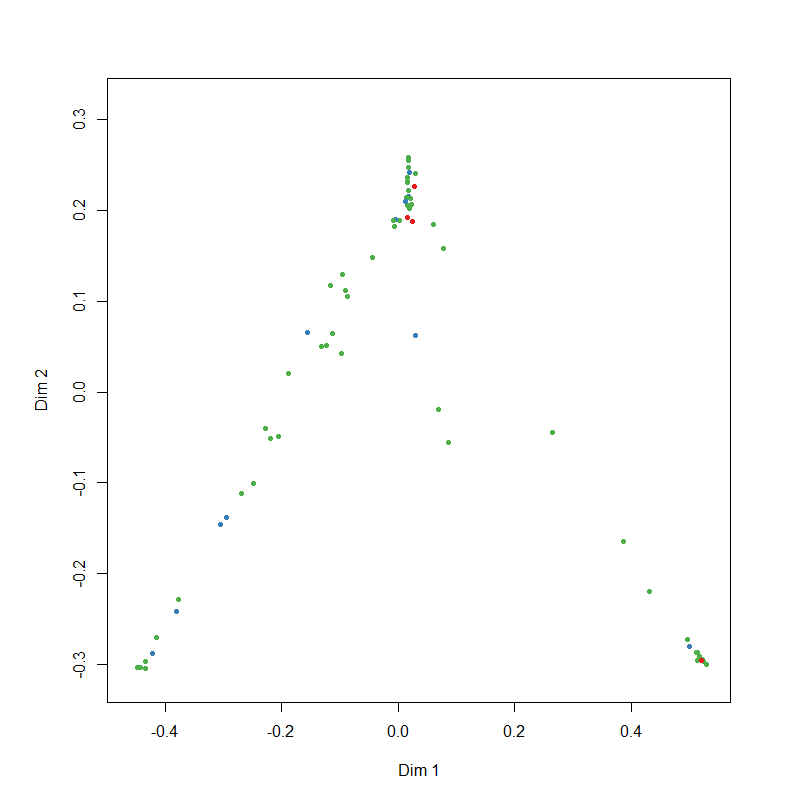
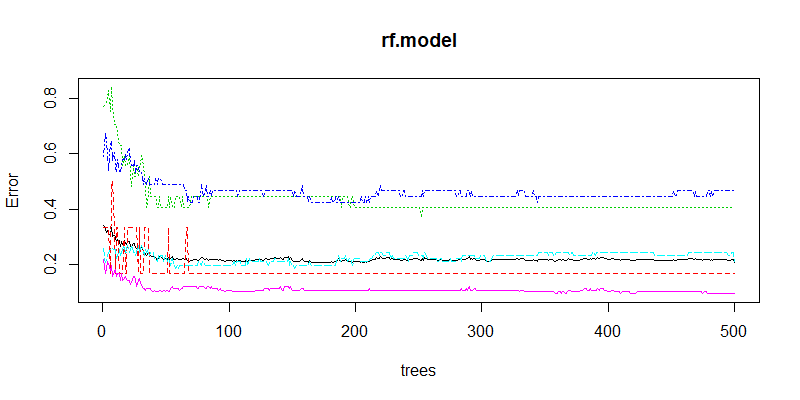
# 説明変数の重要度を表示・プロットする imp <- importance(rf.model, scale = FALSE) print(imp, digits = 4) dotchart(sort(imp [, 7]), xlab = "ジニ係数の平均減少量", ylab = "", main = "各項目の重要度") # 予測変数内の類似度を多次元尺度法で図示 MDSplot(rf.model, dat$IV) # 機械学習の収束状況をプロット plot(rf.model)
最適な特徴量とtreeの数を調べてモデルを再構築
rfTuning <- tuneRF(x = features, y = labels, stepFactor = 2, improve = 0.05, trace = TRUE, plot = TRUE, doBest = TRUE) rf.model2 <- randomForest( x = features, y = labels, mtry = 6, #エラーの最も低いmtry数を指定 ntree = 200, #モデルが収束したntreeを指定 importance = T ) # 分類の正確さの確認 prdct <- predict(rf.model2, newdata = test) table(prdct, test$IV) correctAns <- 0 for (i in 1:nrow (table (prdct, test$IV))) correctAns <- correctAns + table (prdct, test$IV) [i, i] correctAns/nrow (test)